L’analyse d’un prélèvement ADN va débuter par une étape obligatoire d’extraction et de purification. Pour pouvoir analyser la molécule d’ADN il faut en effet la “décrocher” de son support en la mettant dans un milieu aqueux. Des substances présentes sur le prélèvement d’ADN vont se dissoudre par la même occasion. Ces substances qui peuvent interférer avec le processus d’analyse seront éliminées et seules les molécules d’ADN seront conservées. L’étape suivante sera l’amplification par PCR afin d’obtenir une quantité d’ADN suffisante pour l’analyse.
La PCR
La PCR (Polymerase Chain Reaction) est une technique d’amplification qui permet de “photocopier” l’ADN dans une éprouvette. Des parties de la molécule ADN sont sélectionnés par ce qu’on appelle des “amorces” qui sélectionnent la zone à copier. La zone est copiée de la même manière que lors d’une division cellulaire. C’est sous l’action de l’enzyme nommée ADN polymérase que des brins vont se séparer avant de se recombiner avec d’autres nucléotides. Les nucléotides G – C et T – A étant complémentaires la chaîne se reforme à l’identique.
La technique de la PCR s’effectue en trois grandes étapes :
• L’étape de dénaturation : les deux brins sont dissociés par un chauffage à 95°C
• L’étape d’hybridation : la température est abaissée (entre 50 et 60°C) et les amorces viennent se fixer aux parties d’ADN à copier.
• L’étape de polymérisation ou d’élongation : l’ADN polymérase effectue un assemblage de nucléotides sur la chaîne en construction
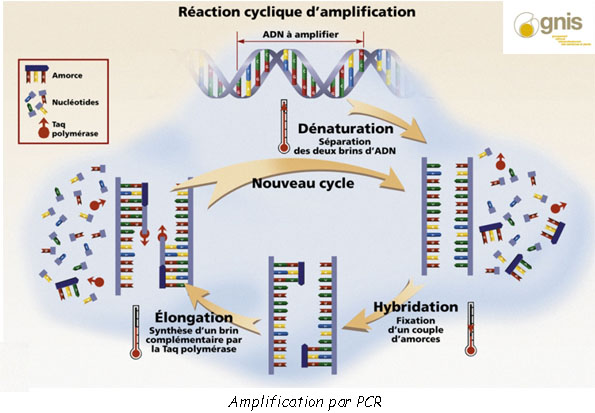
Chaque cycle de copiage permet ainsi de doubler la quantité d’ADN et l’on va très rapidement obtenir des milliards de copies de la zone sélectionnée.
La PCR a ouvert la voie à l’analyse de très petits échantillons contenant quelques cellules. Cependant on considère que pour obtenir sans difficulté un profil de qualité, il est nécessaire d’analyser une centaine de cellules, c’est à dire un quantité d’ADN proche d’un milliardième de gramme (1ng). En deça de cette quantité, des résultats peuvent être faussés par la dégradation du prélèvement ou par des contaminations.
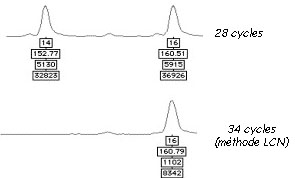 En ce qui concerne les dégradations, lorsque l’analyse porte sur deux à trois molécules d’ADN, la moindre dégradation d’1 à 2 molécules d’ADN peut conduire à une absence de pic après électrophorèse et donc à un profil incomplet voir erroné. Prenons l’exemple d’un prélèvement composé de deux cellules d’un individu possédant les allèles 14 et 16 pour le marqueur D5S818. Si un dommage (mutation) survient sur deux molécules possédant l’allèle 14, on constatera la disparition du pic correspondant. L’analyste pourra alors croire que l’individu est homozygote et possède uniquement l’allèle 16.
En ce qui concerne les dégradations, lorsque l’analyse porte sur deux à trois molécules d’ADN, la moindre dégradation d’1 à 2 molécules d’ADN peut conduire à une absence de pic après électrophorèse et donc à un profil incomplet voir erroné. Prenons l’exemple d’un prélèvement composé de deux cellules d’un individu possédant les allèles 14 et 16 pour le marqueur D5S818. Si un dommage (mutation) survient sur deux molécules possédant l’allèle 14, on constatera la disparition du pic correspondant. L’analyste pourra alors croire que l’individu est homozygote et possède uniquement l’allèle 16.
La PCR et le risque de contamination
Pour les contaminations, le simple fait d’augmenter le nombre de cycles de PCR peut provoquer l’apparition de pics qui ne proviennent pas de l’ADN de l’échantillon mais de pollutions environnantes (opérateur, matériel et environnement de travail, atmosphère). Bien que toutes les précautions soient prises pour éviter ces contaminations, il est difficile d’obtenir un environnement avec une absence totale d’ADN. Lorsque la PCR est poussée à l’extrême les contaminations même infimes sont détectées. Et c’est pour cette même raison que les laborantins qui analysent l’ADN doivent envisager des solution drastiques pour éviter les contaminations (matériel et plans de travail stériles, combinaison intégrale) et que les spécialistes de scène de crime doivent revêtir des vêtements de protection complets (gants, masque, combinaison intégrale). L’absence de ces mesures peut mener à des contaminations de scellés comme cela a été constaté dans l’affaire Treiber ou dans l’affaire de la Tuerie de Chevaline
Malgré ces difficultés des laboratoires se sont spécialisés dans l’analyse de ces échantillons ne contenant que quelques cellules. La méthode LCN (Low Copy Number) a ainsi été développée pour répondre à ces nouvelles problématiques.
L’électrophorèse : dernière étape de l’analyse
Depuis les années 80 et encore aujourd’hui les analyses ADN sont effectuées par électrophorèse. L’électrophorèse est une technique permettant de séparer des molécules en fonction de leur taille en leur appliquant un champ électrique. La molécule ADN étant chargé négativement, les morceaux d’ADN migrent dans un milieu gélatineux (composé de polyacrylamide) sous l’action d’un champ électrique. La migration est d’autant plus rapide que le fragment d’ADN utilisé est court. Le milieu gélatineux, plus stable qu’un liquide classique, joue le rôle de “tamis moléculaire” en gênant la progression des plus grosses molécules. Les premières analyses ADN étaient effectuées sur des supports gélatineux rectangulaires et les résultats ressemblaient à des codes barres
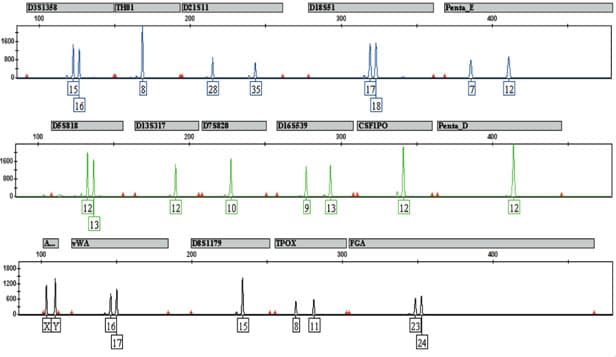
L’électrophorèse capillaire a peu à peu remplacé l’électrophorèse sur polyacrylamide. Le principe est identique a celui d’une électrophorèse classique mais la migration se fait dans un tube de la taille d’un cheveu. Des tensions élevées sont appliquées ce qui permet une séparation rapide des molécules. Un détecteur placé à la sortie du tube enregistre la sortie des morceaux d’ADN en fonction du temps. Les résultats sont donc présentés différemment sous forme de graphique avec en abscisse le temps et en ordonnée la quantité d’ADN détecté (cf ci-dessous)

Les analyses en multiplex
Les techniques d’analyse d’ADN utilisées aujourd’hui permettent d’analyser plusieurs STR en même temps : on parle d’analyse en multiplex.
Des amorces vont sélectionner des zones d’ADN à amplifier par PCR. Plusieurs fragments (jusqu’à environ 15 fragments) sont amplifiés de manière simultanée par une analyse en mode “multiplex”. Les fragments amplifiés sont des STR qui ont été choisis en fonction de leurs polymorphismes et de leur taille.
A la sortie de l’électrophorèse, des fragments d’ADN de différentes tailles sont détectées. Mais si le fragment a une taille de 120 nucléotides comment savoir si celui-ci provient de la répétition de AACG 30 fois (4 nucléotides x 30 = 120 nucléotides) ou alors de la répétition de AACGT 24 fois (5 nucléotides x 24 = 120 nucléotides) ? On peut imaginer la difficulté d’associer l’allèle détecté à un STR particulier. La première des solutions est donc de choisir des STR qui ont des gammes d’allèles bien distinctes. Ainsi le premier STR aura des allèles composé de 64 à 120 nucléotides, le deuxième STR aura des allèles composés de 130 à 186 nucléotides, le troisième STR aura des allèles composés de 200 à 276 nucléotides…. La taille du fragment de sortie permettra automatiquement de l’associer à un STR défini.
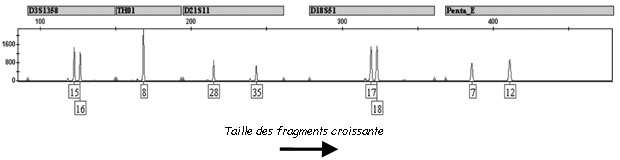
La deuxième solution est de marquer les différents STR par des colorant fluorescents. Les colorants fluorescents sont accrochés aux amorces et les allèles détectés à la sortie sont automatiquement associés au STR. Cette solution peut se combiner à la première ce qui donne le résultat suivant :
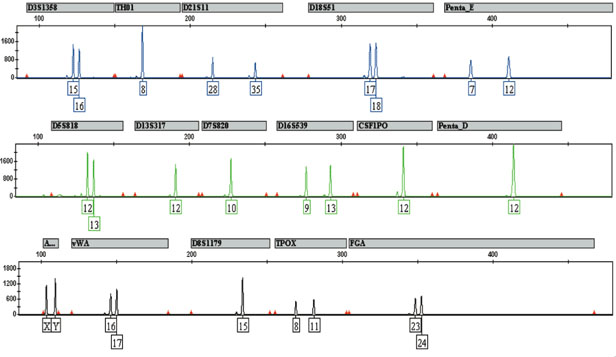
Les Kits multiplex utilisés sont les mêmes dans tous les laboratoires car ce sont des kits “standards” créés par des sociétés de biotechnologie. Outre leur marque commerciale, les kits multiplex différent par les marqueurs qu’ils vont sélectionner. Une partie des marqueurs analysés sont les mêmes dans tous les pays européen ce qui permet un échange des données.
En France c’est le Kit Identifiler de la compagnie ABI qui est utilisé et qui a la vocation d’analyser 15 marqueurs.